Ein chemischer Werkzeugkasten für die Biologie
- D-CHAB
- LOC
- Highlights
Bisweilen muss man etwas Künstliches schaffen, um die Natur zu verstehen. Kathrin Lang, neue Professorin für chemische Biologie am LOC, entwickelt dafür spezielle Werkzeuge und öffnet damit molekulare Türen, die nicht nur Einblicke in bestehende komplexe biologische Systeme gestatten, sondern auch zeigen, was jenseits davon möglich ist.

„Du bisch a aus Südtirol?“ Das Eis ist gebrochen, noch bevor man zwischen den fünf Grünpflanzen in dem Büro Platz genommen hat. Zwei Südtirolerinnen sitzen sich dort am Tisch gegenüber: eine interviewt, die andere, um die es in diesem Text geht, stammt aus Gries/Bozen. Sie heisst Kathrin Lang und zeigt gerade stolz mit beiden Händen, wie viel der grosse Kaktus hinter ihr gewachsen ist, seit man ihn ihr als Abschiedsgeschenk an der TU München überreicht hat. Im Sommer 2021 ist Kathrin Lang als ordentliche Professorin an die ETH Zürich gekommen, mit Doktoranden, Postdocs und etlichen molekularen Werkzeugen im Gepäck. Dieses chemische Repertoire will sie nun an der ETH erweitern, um das Leben der wichtigsten Spieler am biologischen Spielfeld näher zu beleuchten: der Proteine.
„Eigentlich wollte ich immer Mathematik studieren und naturwissenschaftliche Probleme damit lösen“, erinnert sich Kathrin Lang. Das Technomathematik-Studium in München habe sie dennoch abgebrochen, als ihr klar geworden sei, dass sie nicht studiere, um später Zündvorgänge bei BMW zu optimieren. Sie entschied sich für Chemie in Innsbruck, wo sie ihren Platz schliesslich in der chemischen Biologie fand. „Im Doktorat habe ich modifizierte RNA hergestellt und mit Ribosomen gearbeitet. Das sind jene komplexen Maschinen in der Zelle, die anhand von genetischer Information Proteine bauen. Absolut faszinierend“, schwärmt Lang.
Ihr Interesse führte sie 2009 als Postdoc nach Cambridge, zu Venki Ramakrishnan, der just in dem Jahr den Nobelpreis für seine kristallographischen Untersuchungen von Ribosomen erhielt. „Seine hochaufgelösten Strukturbilder lieferten erstmals Einblicke in die Proteinfabriken der Zelle. Das war spannend“, erinnert sich die Chemikerin. Doch die praktische Chemie fehlte ihr. Sie wechselte erst zur Gruppe von Jason Chin in Cambridge, wo sie zum ersten Mal mit synthetischer Biologie in Berührung kam, dann 2014 für eine Professur an die TU München. Seither forscht sie mit ihrem Team im Bereich chemischer und synthetischer Biologie.

"Was ich nicht herstellen kann, kann ich nicht verstehen"
Richard Feynman hat gesagt: „Was ich nicht herstellen kann, kann ich nicht verstehen.“ Dem Grundsatz folgt man auch in der chemischen und synthetischen Biologie. Es ist ein interdisziplinäres Fach im Grenzbereich von organischer Chemie, Biotechnologie, Molekularbiologie und Ingenieurswissenschaften. Das sei das Faszinierende daran, findet Kathrin Lang, die ein Fan von kreativen out-of-the-box-Ansätzen ist. „In meiner Gruppe entwickeln wir chemische Werkzeuge, um biologische Systeme zu erzeugen, die in der Natur so nicht vorkommen. Wir bauen sie nach und modifizieren sie. Auf die Art können wir biologische Fragestellungen beantworten – etwa warum die Natur etwas so macht und nicht anders. Gleichzeitig können wir Dinge schaffen, die in der Natur nicht möglich sind.“
Das Anwendungsfeld dafür ist breit. So lassen sich etwa Bakterien modifizieren, damit sie Plastik abbauen können, Enzyme generieren, die neue chemische Reaktionen ausführen oder Antikörper herstellen, die bestimmte Krebszellen angreifen. Inspiration für ihre Tools holt sich Kathrin Lang im Austausch mit biologischen Instituten oder auf biologischen Symposien. Sie denkt dabei noch immer chemisch, in Atomen, Molekülen und Mechanismen, selbst wenn es um biologische Prozesse geht. Drei biologische Fragestellungen beschäftigen die Lang-Arbeitsgruppe derzeit besonders und bilden die Säulen der Forschung an der ETH Zürich: wie kann man Proteine mit biophysikalischen Sonden versehen, sie z.B .sichtbar machen? Wann und wie interagieren Proteine mit anderen Proteinen in der Zelle? und warum und wie werden Proteine nach ihrer Synthese modifiziert, etwa ubiquitiniert?
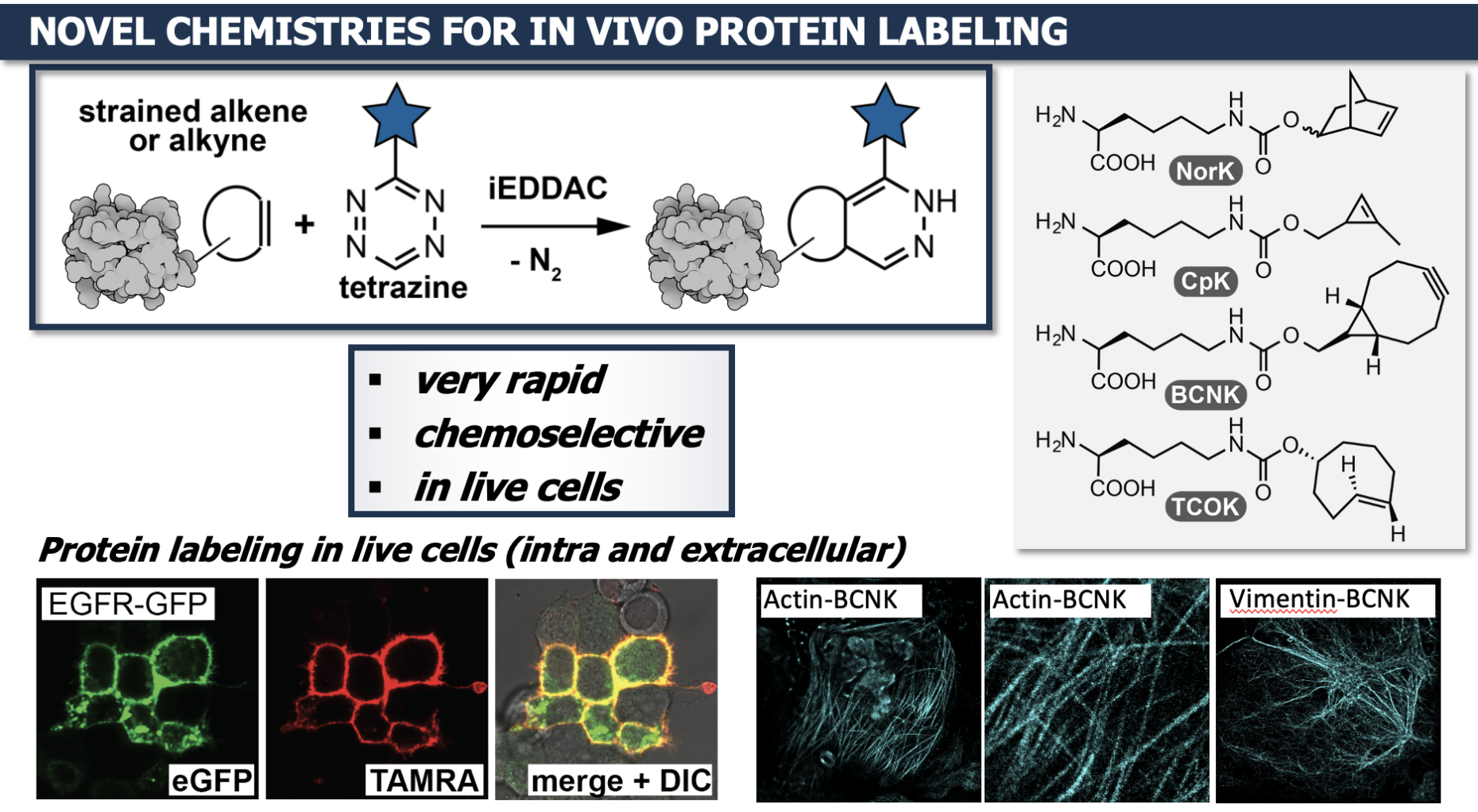
Jenseits der 20 natürlichen Aminosäuren
„Jedes Lebeweisen, vom einfachsten Einzeller bis zu uns Menschen, verwendet die gleichen 20 Aminosäuren. Sie sind die Bausteine für unsere Proteine und die Reihenfolge dieser 20 Bausteine ist im genetischen Code festgelegt“, erklärt Kathrin Lang. „Meine Gruppe arbeitet nun daran, den hoch-konservierten genetischen Code zu erweitern, um artifizielle Aminosäuren jenseits dieser 20 in Proteine in lebenden Zellen einzubauen. Dies ermöglicht es Proteine mit neuen funktionellen Gruppen und mit neuen physikalisch-chemischen Eigenschaften auszustatten.“
Besonders interessiert man sich im Labor von Kathrin Lang fürs Labeling, das heisst in dem Fall für das Sichtbarmachen von Proteinen. Bislang genutzte Fluoreszenzproteine wie GFP sind oft gross und können das Zielprotein beeinflussen. Stattdessen arbeitet man auch hier mit modifizierten Aminosäuren. „Diese sind mit einer Art Henkel funktionalisiert. Dieser Henkel soll dann in der lebenden Zelle mit seinem ‚Gegenstück‘, welches einen fluoreszierenden Farbstoff, ein Fluorophor, trägt, reagieren und man erhält ein fluoreszierendes Protein“, erklärt Lang. „Man könnte damit beispielsweise in Krebszellen verfolgen, was mit dem Protein passiert. Wird es vielleicht in ein anderes Kompartment der Zelle transportiert?“ Die Entwicklung von solchen Reaktionen ist sehr komplex, denn die Reaktion zwischen Henkel und Fluorophor muss ausserordentlich selektiv sein und dies in der denkbar komplexesten Umgebung: in der lebenden Zelle. Da könne man nicht eben mal aufheizen oder toxische Mittel anwenden, wenn eine Reaktion nicht funktioniert. „Unsere Tools müssen unbeeinflusst von allem funktionieren, was in den Zellen sonst noch an Chemie passiert, und das ist viel.“ Idealerweise könne man künftig auch mehrere verschiedene Fluorophore einbringen. Damit könnte man studieren, wie sich Proteine im zellulären Milieu falten, aber da arbeite man noch dran.
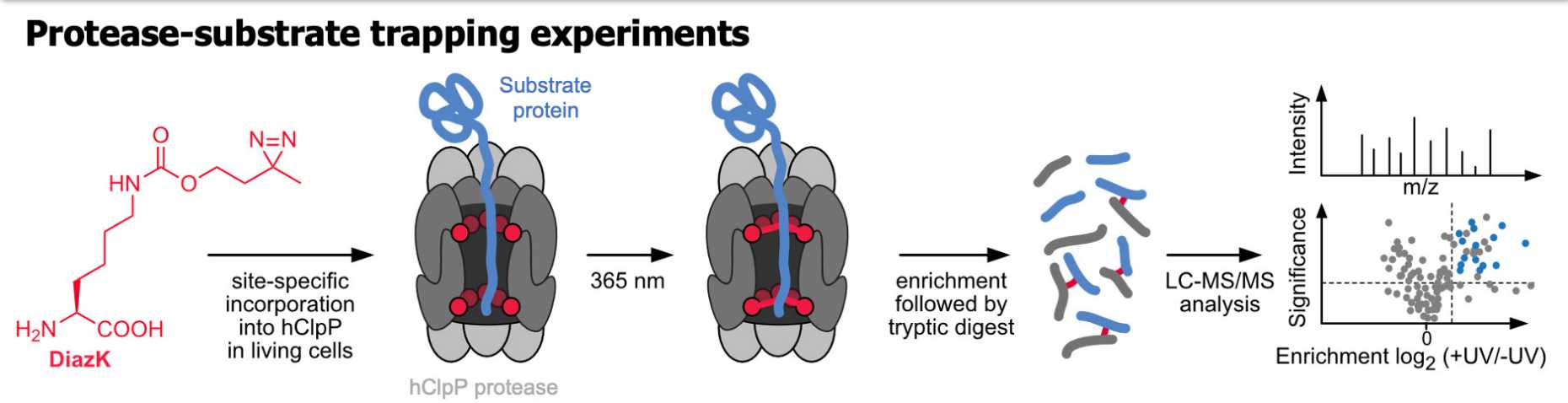
Wichtig zu verstehen ist auch, dass Proteine nicht in Isolation agieren, sondern in ihrer natürlichen Umgebung verschiedenste Interaktionen eingehen. Um dies zu studieren, hat Kathrin Langs Gruppe modifizierte Aminosäuren entwickelt, die in ein Protein eingebaut werden können und bei Anregung mit Licht eine kovalente Bindung mit einem Protein in der Nähe eingehen. „In Mitochondrien gibt es zum Beispiel ein Abbausystem für Proteine, von dem man zwar wusste, dass es bei bestimmten Krankheiten wichtig ist; man kannte aber die Proteine nicht, die darin abgebaut werden“, erzählt Lang. „Wir haben also unsere Aminosäuren in diesem System positioniert. Dadurch konnten die vorbeiwandernden Proteine ‚eingefangen‘ und am Ende identifiziert werden.“
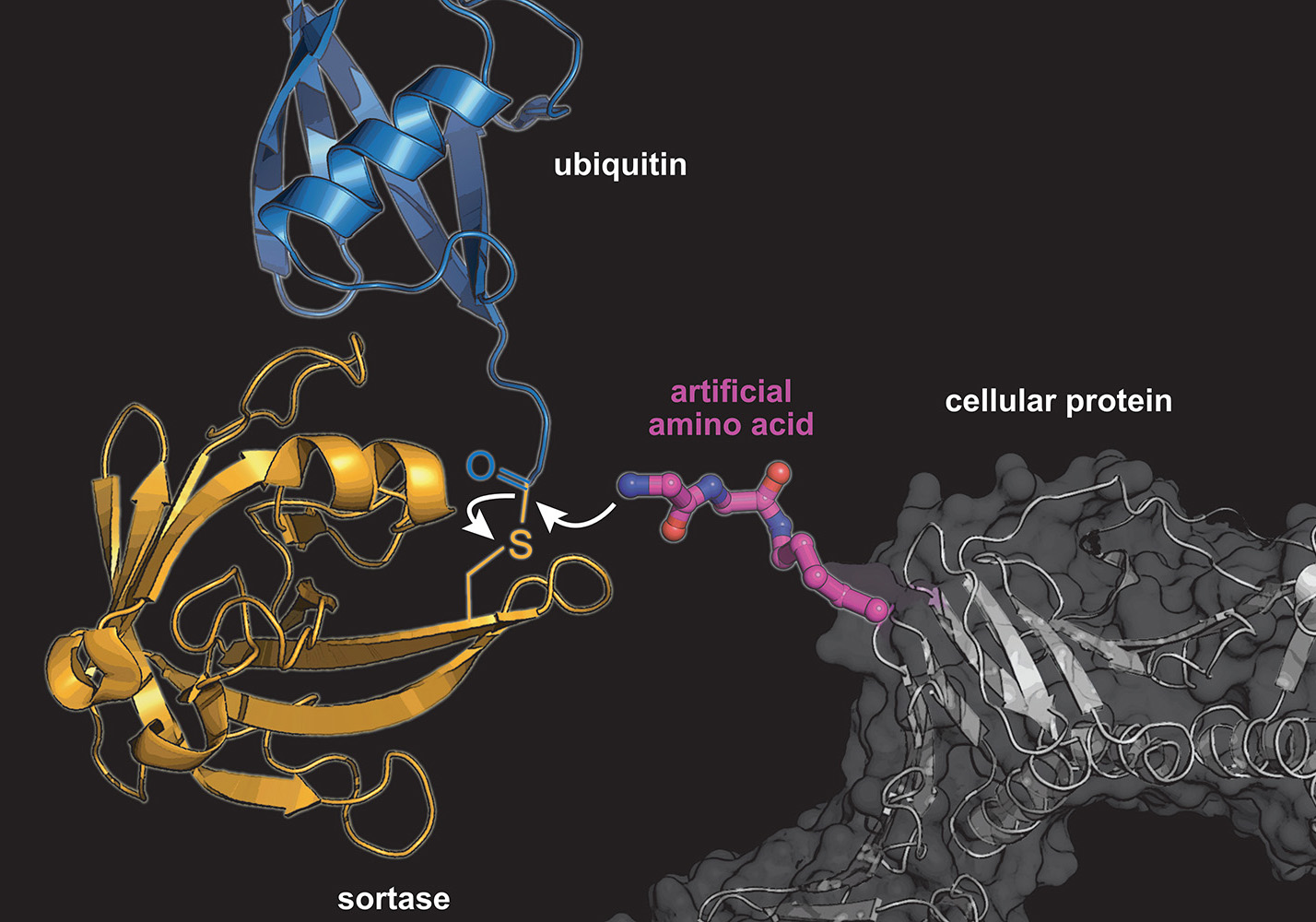
Ein besonders wichtiger biologischer Regulationmechanismus besteht in der Modifikation von Proteinen nach deren Biosynthese. Dazu gehört z.B. die Ubiquitinierung, bei welcher eines oder mehrere kleine Ubiquitinproteine an ein Zielprotein gehängt werden. Art, Position und Anzahl der an ein Protein gebundenen Ubiquitin-Moleküle bestimmen Stabilität, Funktion und Aufenthaltsort des Proteins innerhalb der Zelle. „Praktisch jeder zelluläre Prozess wird durch Ubiquitinierung beeinflusst. Fehlfunktionen im Ubiquitinierungsmechanismus führen zu vielen schweren Krankheiten wie etwa Krebs“, erklärt Kathrin Lang. Aufgrund der Komplexität der Ubiquitinierung, ist es aber schwierig, in solche Systeme hineinzuschauen. Daher entwickeln wir Methoden, um diese komplexen System nachzubauen – sowohl im Reagenzglas als auch in der lebenden Zelle. Dies erlaubt es uns dann die Auswirkungen von Ubiquitin-Markierungen beispielsweise auf das Entstehen von Krebs oder neurogenerativen Erkrankungen auf molekularer Ebene zu studieren.
Vielseitig anwendbare Werkzeuge
„Der Wert eines Werkzeugs ergibt sich durch seine breite Anwendbarkeit“, betont Lang, „Unser Ziel ist es, dass auch Zellbiologen, Neurobiologen oder Biochemiker unsere Sachen nachbauen und nutzen können und dass unsere Tools es ermöglichen, biologische Prozesse zu entschlüsseln, die mit traditionelleren Herangehensweisen nicht studiert werden können. Wir haben auch schon einige Dinge patentieren lassen, und arbeiten daran, dass viele unserer Werkzeuge auch irgendwann als Kit mit einfacher Anleitung zur Verwendung in jedem biologischen Labor verwendet werden können."
Fürs Erste gilt es nun die Laborgruppe weiter auszubauen – und zwar so, dass sich alle gut verstehen und die Chemie passt, arbeitet man hier doch stark interdisziplinär und begrüsst neue thematische Herausforderungen im Grenzgebiet zwischen Chemie und Biologie. Ausserdem müsse sie jetzt – nach Corona und Umzugsstress – endlich mal auf ein paar Schweizer Berge steigen und den Bekanntenkreis in Zürich erweitern, ergänzt Kathrin Lang lachend. Das Interview mit der Südtirolerin, die nun am anderen Ende des Tisches ihr Aufnahmegerät einpackt, war schon mal ein guter Start dafür.

Einführungsvorlesung - Kathrin Lang
An expanded genetic code – adding new chemistries to biology
1. Juni 2022
Zeit: 17.15 – 18.30
Ort: Zürich Zentrum HG F 30
Livestream: https://video.ethz.ch/live/events/live-1.html
Gruppenwebsite
Literatur und weiterführende Informationen
Kathrin Lang, Lloyd Davis, Stephen Wallace, Mohan Mahesh, Daniel J. Cox, Melissa L. Blackman, Joseph M. Fox, and Jason W. Chin (2012): Genetic Encoding of Bicyclononynes and trans-Cyclooctenes for Site-Specific Protein Labeling in Vitro and in Live Mammalian Cells via Rapid Fluorogenic Diels–Alder Reactions. Journal of the American Chemical Society 2012 134 (25), 10317-10320. doi: externe Seite10.1021/ja302832gcall_made
Chayasith Uttamapinant, Jonathan D. Howe, Kathrin Lang, Václav Beránek, Lloyd Davis, Mohan Mahesh, Nicholas P. Barry, and Jason W. Chin (2015): Genetic Code Expansion Enables Live-Cell and Super-Resolution Imaging of Site-Specifically Labeled Cellular Proteins. Journal of the American Chemical Society 2015 137 (14), 4602-4605 doi: externe Seite10.1021/ja512838zcall_made
Nguyen T. A. , Gronauer T. F., Nast-Kolb T. , Sieber S.A , Lang K. (2021): Substrate Profiling of Mitochondrial Caseinolytic Protease P via a Site-Specific Photocrosslinking Approach. Angew. Chem.Int. 2022,61,e202111085. doi: externe Seite10.1002/anie.202111085call_made
S.V. Mayer, A. Murnauer, M.K. von Wrisberg, M.L. Jokisch and K. Lang (2019): Photo-Induced and Rapid Labeling of Tetrazine-Bearing Proteins via Cyclopropenone-Caged Bicyclononynes. Angew. Chem. Int, 58, 15876-15882 doi: externe Seite10.1002/anie.201908209call_made
M. Fottner, A-D. Brunner, V. Bittl, D. Horn-Ghetko, A. Jussupow, V.R.I. Kaila, A. Bremm and K. Lang (2019): Site-specific ubiquitylation and SUMOylation using genetic code expansion and sortase. Nat. Chem. Biol. , 15, 276-284 doi: externe Seite10.1038/s41589-019-0227-4call_made
T.A. Nguyen, M. Cigler, K. Lang. (2018): Expanding the Genetic Code to Study Protein–Protein Interactions Angew. Chem. Int. Ed. 2018, 57, 14350-14361 doi: externe Seite10.1002/anie.201805869call_made
